Моделі сіамських нейронних мереж для класифікації серцевих аритмій за умов нестачі тренувальних ЕКГ сигналів
Основний зміст сторінки статті
Анотація
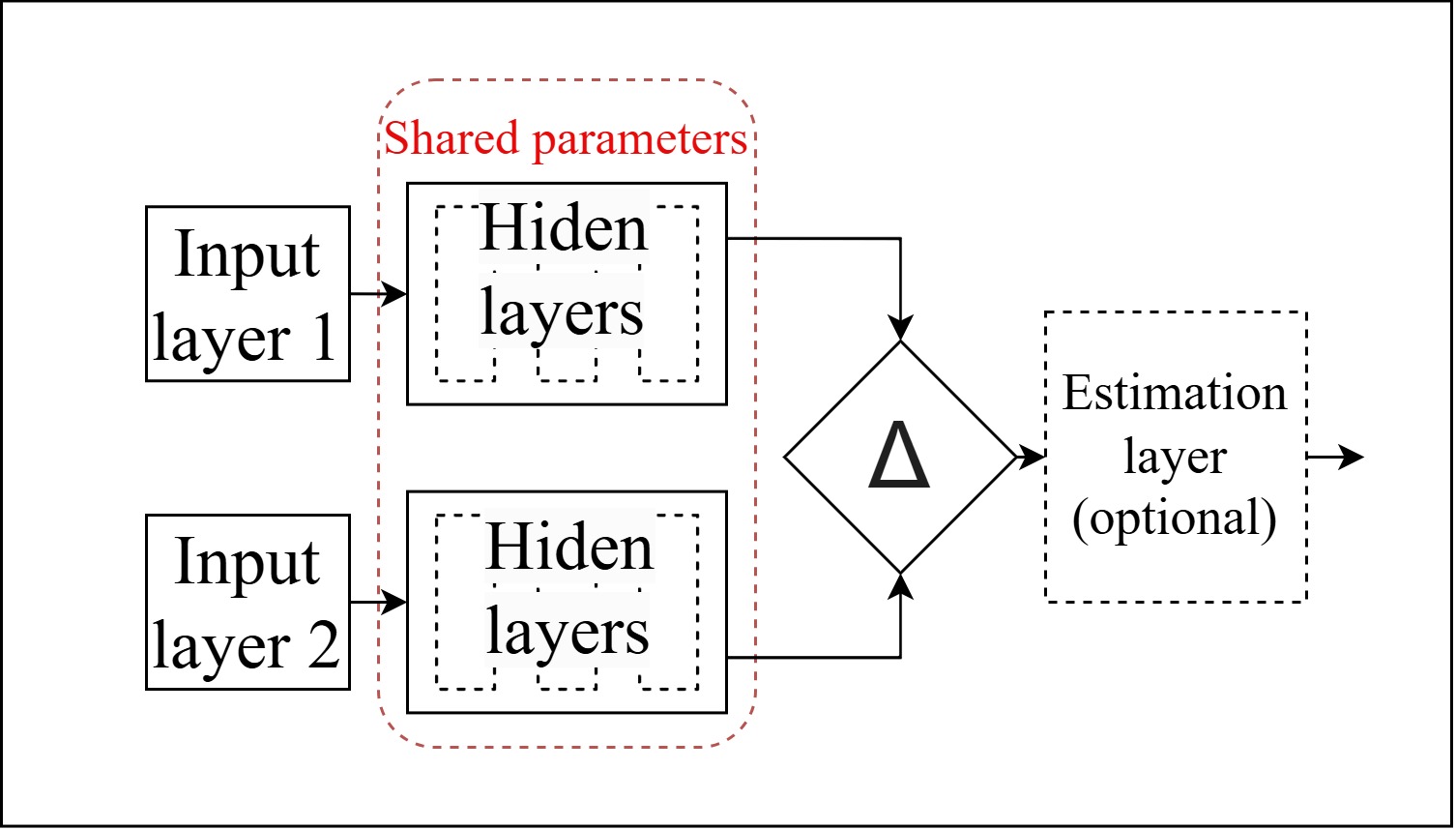
Стаття присвячена створенню моделей сіамських нейронних мереж для класифікації ЕКГ-сигналів, що відображають серцево-судинні патології, зокрема аритмії, в умовах обмеженої кількості тренувальних даних. Проблема дефіциту навчальних зразків у машинному навчанні для діагностики серцевих захворювань пов’язана з великою різноманітністю патологічних станів і недостатньою інформацією для окремих класів у відкритих медичних базах даних. Дослідження спрямоване на розробку комплексного методу, який ґрунтується на поєднанні методів електрокардіографії високого розрізнення та векторкардіографії з архітектурами та методами навчання сіамських нейронних мереж, що дає можливість підвищити точність класифікації серцевих аритмій. Особливість запропонованого метода, яка базується на здатності сіамських НМ до порівняння, полягає у виявленні та аналізі відмінностей між ЕКГ сигналом, що досліджується, та сформованим еталонним вектором ознак сигналів з патологією, що дозволяє ефективно ідентифікувати зміни сигналів навіть для тих захворювань, які обмежено-представлені в навчальному наборі даних. Крім того, для підвищення ефективності навчання був розроблений метод формування еталонного вхідного вектора ознак захворювання, який використовується сіамською нейронною мережею для порівняння. Застосування методу головних компонент (PCA) дозволило виділити ключові ознаки зі 100 ЕКГ-сигналів із патологіями, що сприяло створенню еталонного вектора ознак із мінімальною кількістю тренувальних зразків. Додатково для кожного вхідного ЕКГ-сигналу та еталонного вектора розраховувався усереднений кардіоцикл, що сприяло ідентифікації низькоамплітудних компонентів ЕКГ та особливостей QRS комплексу. Для реалізації розробленого комплексного методу використовувалася база PTB-XL, що містить 12-канальні ЕКГ-записи, класифіковані за 70 категоріями захворювань. Для зменшення впливу дисбалансу даних застосовано методи аугментації, а також методи попередньої обробки шляхом видалення зашумлених сигналів та вибіркове скорочення надмірно представлених класів. В рамках дослідження розроблено дві моделі сіамських нейронних мереж. Перша модель орієнтована на виявлення низькоамплітудних патологічних компонент ЕКГ сигналів, зокрема пізніх потенціалів передсердь та шлуночків. Друга модель, призначена для класифікації 18 типів аритмій і 19 супутніх патологій, таких як ішемічна хвороба серця, гіпертрофія та інфаркт міокарда. Ефективність запропонованих моделей НМ була оцінена шляхом порівняння з мережею «ECGnet» у задачі розпізнавання пізніх потенціалів передсердь і шлуночків. Перша модель перевищила точність «ECGnet» у середньому на 10% та зменшила ймовірність хибнонегативних прогнозів. Друга модель НМ для багатокласової класифікації, яка охоплювала 37 діагностичних класів з рідкісними захворюваннями, що мають менше, ніж 200 спостережень, перевищила середню точність «ECGnet» на 10%, досягаючи максимального приросту в 28%. Отримані результати дозволяють окреслити подальші шляхи вдосконалення комплексного методу. Зокрема, підвищення точності класифікації ЕКГ сигналів з патологіями можливе шляхом використання додаткових перетворень вхідних ознак та методів підсилення низькоамплітудних компонент сигналу.
Блок інформації про статтю

Ця робота ліцензується відповідно до Creative Commons Attribution-ShareAlike 4.0 International License.
Автори, які публікуються у цьому журналі, погоджуються з наступними умовами:- Автори залишають за собою право на авторство своєї роботи та передають журналу право першої публікації цієї роботи на умовах ліцензії Creative Commons Attribution License, котра дозволяє іншим особам вільно розповсюджувати опубліковану роботу з обов'язковим посиланням на авторів оригінальної роботи та першу публікацію роботи у цьому журналі.
- Автори мають право укладати самостійні додаткові угоди щодо неексклюзивного розповсюдження роботи у тому вигляді, в якому вона була опублікована цим журналом (наприклад, розміщувати роботу в електронному сховищі установи або публікувати у складі монографії), за умови збереження посилання на першу публікацію роботи у цьому журналі.
- Політика журналу дозволяє і заохочує розміщення авторами в мережі Інтернет (наприклад, у сховищах установ або на особистих веб-сайтах) рукопису роботи, як до подання цього рукопису до редакції, так і під час його редакційного опрацювання, оскільки це сприяє виникненню продуктивної наукової дискусії та позитивно позначається на оперативності та динаміці цитування опублікованої роботи (див. The Effect of Open Access).
Посилання
D. K. Shreyas, N. J. Srivatsa, K. H. Vishwas, V. Vishaka, and C. S. Kaliprasad, “A Review on Neural Networks and its Applications”, Journal of Computer Technology & Applications, Jan. 2023. DOI: 10.37591/jocta.v14i2.1062
C. F. G. D. Santos and J. P. Papa, “Avoiding Overfitting: A Survey on Regularization Methods for Convolutional Neural Networks”, ACM Computing Surveys, vol. 54, no. 10s, pp. 1–25, Jan. 2022. DOI: 10.1145/3510413.
R. Smisek, A. Nemcova, L. Marsanova, L. Smital, M. Vítek, and J. Kozumplik, “Cardiac Pathologies Detection and Classification in 12-lead ECG”, in 2020 Computing in Cardiology Conference, 2020. DOI: 10.22489/CinC.2020.171.
M. Alhaskir, “ECG Matching: An Approach to Synchronize ECG Datasets for Data Quality Comparisons”, in German Medical Data Sciences 2023 – Science. Close to People.: Proceedings of the 68th Annual Meeting of the German Association of Medical Informatics, Biometry, and Epidemiology e.V. (gmds) 2023 in Heilbronn, Germany, IOS Press, 2023. URL: https://ebooks.iospress.nl/doi/10.3233/SHTI230718.
C. Li, “DeepSpeed Data Efficiency: Improving Deep Learning Model Quality and Training Efficiency via Efficient Data Sampling and Routing”, Proceedings of the AAAI Conference on Artificial Intelligence, vol. 38, no. 16, pp. 18490–18498, Mar. 2024. DOI: 10.1609/aaai.v38i16.29810.
M. Al-Shedivat, L. Li, E. Xing, and A. Talwalkar, 01-Jan.-2021. DOI: 10.48550/arXiv.2102.00127.
S. Narayanaswamy, “High resolution electrocardiography”., Indian Pacing Electrophysiol J, vol. 2, no. 2, pp. 50–56, Apr. 2002. PMID: 17006557.
Q. Chen, S. Kastratovic, M. Eid, and S. Ha, “A Non-Contact Compact Portable ECG Monitoring System”, Electronics, vol. 10, no. 18, p. 2279, Sep. 2021. DOI: 10.3390/electronics10182279.
J. A. AYENI, “Convolutional Neural Network (CNN): The architecture and applications”, Applied Journal of Physical Science, vol. 4, no. 4, pp. 42–50, Dec. 2022. DOI: 10.31248/AJPS2022.085.
I. D. Mienye, T. G. Swart, and G. Obaido, “Recurrent Neural Networks: A Comprehensive Review of Architectures, Variants, and Applications”, Information, vol. 15, no. 9, p. 517, Aug. 2024. DOI: 10.3390/info15090517.
I. Amaya-Rodriguez, “ResNet”, in Computer-Aided Analysis of Gastrointestinal Videos, Cham: Springer International Publishing, 2021, pp. 99–114. URL: https://link.springer.com/10.1007/978-3-030-64340-9_12.
J. Cai, W. Sun, J. Guan, and I. You, “Multi-ECGNet for ECG Arrythmia Multi-Label Classification”, IEEE Access, vol. 8, pp. 110848–110858, Jan. 2020. DOI: 10.1109/ACCESS.2020.3001284.
P. Behrouzi, B. Shirkani, and M. Hazratifard, “Using ECG Signals in Siamese Networks for Authentication in Digital Healthcare Systems”, Journal of Biomedical Research & Environmental Sciences, vol. 3, no. 12, pp. 1367–1373, Nov. 2022. DOI: 10.37871/jbres1605.
T. Schlagenhauf, Y. Lin, and B. Noack, “Discriminative feature learning through feature distance loss”, Machine Vision and Applications, vol. 34, no. 2, Feb. 2023. DOI: 10.1007/s00138-023-01379-1.
M. M. Al Rahhal, Y. Bazi, M. Al Zuair, E. Othman, and B. BenJdira, “Convolutional Neural Networks for Electrocardiogram Classification”, Journal of Medical and Biological Engineering, vol. 38, no. 6, pp. 1014–1025, Mar. 2018. DOI: 10.1007/s40846-018-0389-7.
P. Wagner, “PTB-XL, a large publicly available electrocardiography dataset”, Scientific Data, vol. 7, no. 1, May 2020. DOI: 10.1038/s41597-020-0495-6.
S. Khurshid, “Frequency of Cardiac Rhythm Abnormalities in a Half Million Adults”, Circulation: Arrhythmia and Electrophysiology, vol. 11, no. 7, Jul. 2018. DOI: 10.1161/CIRCEP.118.006273.
X. Fagan, K. Ivanko, and N. Ivanushkina, “Detection of Ventricular Late Potentials in Electrocardiograms Using Machine Learning”, in Advances in Computer Science for Engineering and Education III, Cham: Springer International Publishing, 2020, pp. 487–497. DOI: 10.1007/978-3-030-55506-1_44
M. M. Rahman, M. W. Rivolta, F. Badilini, and R. Sassi, “A Systematic Survey of Data Augmentation of ECG Signals for AI Applications”, Sensors, vol. 23, no. 11, p. 5237, May 2023. DOI: 10.3390/s23115237.
N. Ivanushkina and A. Mnevets, “Detection of Erroneously Selected Cardiac Cycles Using Neural Networks”, in 2024 IEEE 42nd International Conference on Electronics and Nanotechnology (ELNANO), Kyiv, Ukraine, 2024, pp. 406–411. DOI: 10.1109/ELNANO63394.2024.10756903.
A. Doboli and E. H. Currie, “Analog and Digital Filters”, in Introduction to Mixed-Signal, Embedded Design, New York, NY: Springer New York, 2010, pp. 325–371. URL: http://link.springer.com/10.1007/978-1-4419-7446-4_8.
M. Krichane, “Zero Phase Filtering for Active Compensation of Periodic Physiological Motion”, in The First IEEE/RAS-EMBS International Conference on Biomedical Robotics and Biomechatronics, 2006. BioRob 2006., Pisa, Italy, 2006, pp. 182–187. DOI: 10.1109/BIOROB.2006.1639081.
S. Man, A. C. Maan, M. J. Schalij, and C. A. Swenne, “Vectorcardiographic diagnostic & prognostic information derived from the 12‐lead electrocardiogram: Historical review and clinical perspective”, Journal of Electrocardiology, vol. 48, no. 4, pp. 463–475, Jul. 2015. DOI: 10.1016/j.jelectrocard.2015.05.002.
S. Maheshwari, A. Acharyya, M. Schiariti, and P. E. Puddu, “Frank vectorcardiographic system from standard 12 lead ECG: An effort to enhance cardiovascular diagnosis”, Journal of Electrocardiology, vol. 49, no. 2, pp. 231–242, Mar. 2016. DOI: 10.1016/j.jelectrocard.2015.12.008.
R. Jaros, R. Martinek, and L. Danys, “Comparison of Different Electrocardiography with Vectorcardiography Transformations”, Sensors, vol. 19, no. 14, p. 3072, Jul. 2019. DOI: 10.3390/s19143072.
J. Vondrak and M. Penhaker, “Review of Processing Pathological Vectorcardiographic Records for the Detection of Heart Disease”, Frontiers in Physiology, vol. 13, Mar. 2022. DOI: 10.3389/fphys.2022.856590.
M. Yang, “Normalizing Electrocardiograms of Both Healthy Persons and Cardiovascular Disease Patients for Biometric Authentication”, PLoS ONE, vol. 8, no. 8, p. e71523, Aug. 2013. DOI: 10.1371/journal.pone.0071523.
E. J. Berbari and P. Lander, “The methods of recording and analysis of the signal averaged ECG”, in Signal Averaged Electrocardiography, Dordrecht: Springer Netherlands, 1993, pp. 49–68. URL: http://link.springer.com/10.1007/978-94-011-0894-2_4.
F. Castells, P. Laguna, L. Sörnmo, A. Bollmann, and J. M. Roig, “Principal Component Analysis in ECG Signal Processing”, EURASIP Journal on Advances in Signal Processing, vol. 2007, no. 1, Feb. 2007. DOI: 10.1155/2007/74580.